© Universität Bielefeld
uni.aktuell-Archiv
Veröffentlicht am
21. Dezember 2012
Kategorie:
Forschung & Wissenschaft
Ein Puzzle aus Milliarden von Teilen
Bielefelder Centrum für Biotechnologie und Joint Genome Institute,
USA, entschlüsseln Erbinformation von Mikroben in Biogasanlagen
In Biogasanlagen arbeiten Heerscharen von Mikroorganismen täglich daran, aus Abfall Energie herzustellen. Um herauszufinden, welche Mikroben diese Arbeit am besten machen, schauen Forscher vom Centrum für Biotechnologie (CeBiTec) der Universität Bielefeld ganz genau hin: Für ausgewählte Biogasanlagen deutschlandweit analysieren sie die Gesamt-Erbinformation der mikrobiellen Gemeinschaften. Die dazu nötigen Sequenzierarbeiten übernimmt ab Anfang 2013 das kalifornische Joint Genome Institute. Am CeBiTec erfolgt die bioinformatische Analyse. Keine einfache Aufgabe, denn die Daten werden in Milliarden von Bruchstücken geliefert, die wiederum von Hunderten von Organismen stammen. Eine Puzzle-Arbeit.
 In Deutschland gibt es mehr als 7.000 Biogas-Anlagen, die über sechs
Millionen Haushalte mit Strom versorgen können. Die Anlagen werden meist
mit pflanzlicher Biomasse wie Maissilage aber auch mit
landwirtschaftlichen Reststoffen wie Gülle und Hühnerkot befüllt. Eine
der zentralen Forschungsfragen ist, wie sich die Biogas-Produktion
optimieren lässt. Dafür wollen die Bielefelder Wissenschaftler Dr.
Alexander Sczyrba, Dr. Andreas Schlüter, Dr. Alexander Goesmann,
Professor Dr. Jens Stoye und Professor Dr. Alfred Pühler wissen, welche
Mikroben für den Abbau der Biomasse verantwortlich sind – und welche
dies am besten können. „Uns interessiert, welche Mikrobiologie konkret
hinter den Abläufen in der Biogasanlage steckt, welche Mikroorganismen
in welchem Stadium welche Rolle spielen", erklärt Andreas Schlüter, der
am CeBiTec auf dem Gebiet der Biogas-Erzeugung forscht.
In Deutschland gibt es mehr als 7.000 Biogas-Anlagen, die über sechs
Millionen Haushalte mit Strom versorgen können. Die Anlagen werden meist
mit pflanzlicher Biomasse wie Maissilage aber auch mit
landwirtschaftlichen Reststoffen wie Gülle und Hühnerkot befüllt. Eine
der zentralen Forschungsfragen ist, wie sich die Biogas-Produktion
optimieren lässt. Dafür wollen die Bielefelder Wissenschaftler Dr.
Alexander Sczyrba, Dr. Andreas Schlüter, Dr. Alexander Goesmann,
Professor Dr. Jens Stoye und Professor Dr. Alfred Pühler wissen, welche
Mikroben für den Abbau der Biomasse verantwortlich sind – und welche
dies am besten können. „Uns interessiert, welche Mikrobiologie konkret
hinter den Abläufen in der Biogasanlage steckt, welche Mikroorganismen
in welchem Stadium welche Rolle spielen", erklärt Andreas Schlüter, der
am CeBiTec auf dem Gebiet der Biogas-Erzeugung forscht.
Erstes Genom entschlüsselt
Einen ersten Erfolg können die Forscher bereits verbuchen. „Die Genomsequenz von einem Methanproduzenten, Methanoculleus bourgensis, konnte am CeBiTec komplett entschlüsselt werden“, berichtet Professor Pühler. Damit wurde in Bielefeld die erste Genomsequenz für ein Methan-produzierendes Archaeon aus einer Biogasanlagen erstellt – ein einzelliges Urbakterium, das in bestimmten Biogas-Anlagen eine wichtige Rolle spielt. Nun wollen die Forscher mehr.
 Das Puzzle zusammensetzen
Das Puzzle zusammensetzen
Das Projekt ist Teil des „Community Sequencing Programs“, eines öffentlichen Sequenzierungsprogramms, das am Joint Genome Institute vom amerikanischen Energieministerium finanziert wird. Während bisherige Biogas-Studien sich hauptsächlich auf bestimmte Markergene konzentriert haben, soll jetzt die gesamte Erbinformation der Mikroorganismen untersucht werden. Dazu wird das amerikanische Institut mehr als ein Terabyte an Sequenz-Daten produzieren, was etwa dem Umfang von 300 menschlichen Genomen entspricht. Allerdings werden diese Daten in unzähligen Bruchstücken geliefert. Denn die Millionen von Basen, aus denen ein mikrobielles DNA-Molekül besteht, lassen sich auch mit modernster Technik nicht an einem Stück lesen. Stattdessen liefern die Sequenziertechnologien sich überlappende Abschnitte von etwa 150 Basen - davon aber riesige Mengen. In Milliarden von Bruchstücken werden die DNA-Sequenzen wieder nach Bielefeld geschickt, wo die Arbeitsgruppe „Computational Metagenomics“ (Computergestützte Metagenomik) von Alexander Sczyrba ins Spiel kommt. Sie entwickelt bioinformatische Verfahren zur Rekonstruktion von Genomsequenzen. Ihre Aufgabe ist es, die Daten zu vergleichen, die Überlappungen festzustellen und die Basenabfolge anhand dieser „Klebestellen“ wieder zusammenzusetzen. „Wir versuchen ein Puzzle zu lösen, das aus Milliarden von Teilen besteht, wobei auch noch Hunderte von verschiedenen Puzzle-Spielen miteinander vermischt wurden“, erläutert Sczyrba.
Einzelzell-Genomik verspricht neue Erkenntnisse
Ganz nebenbei werden die Bielefelder Forscher neue Wege in der Genomik beschreiten. Schätzungsweise 99 Prozent aller Mikroorganismen lassen sich im Labor nicht kultivieren. Hier soll eine ganz neue Technologie, die Einzelzell-Genomik, Erkenntnisse bringen, indem die Genomsequenz von einzelnen mikrobiellen Zellen bestimmt wird. Es wird erwartet, dass Kenntnisse über Identität und Funktionen bisher komplett unbekannter Mikroorganismen gewonnen werden können. Im Rahmen des Gemeinschaftsprojekts wird das Joint Genome Institute etwa 100 Einzelzell-Genome sequenzieren.
Rund zwei Jahre haben die Forscher für ihr Projekt eingeplant, an dem auch Bielefelder Doktorandinnen und Doktoranden des Graduiertenclusters in Industrieller Biotechnologie (CLIB) beteiligt sind. Am Ende hoffen sie, die optimale Mikroben-Gemeinschaft für Biogasanlagen zu kennen – und damit dieses Verfahren der Energiegewinnung noch effizienter gestalten zu können.
Hintergrund
Biogasanlagen erzeugen durch die Vergärung von pflanzlicher Biomasse Methan, das für Strom- und Wärmeerzeugung genutzt werden kann. Den Abbau der pflanzlichen Biomasse und die Produktion des Biogases in den landwirtschaftlichen Biogasanlagen wird durch Mikroben bewerkstelligt. Dieser Prozess, der in ähnlicher Form auch im Verdauungstrakt von Rindern abläuft, weist eine ausgeglichene Kohlendioxid-Bilanz auf und trägt damit nicht zum “Treibhauseffekt“ bei. Im Gegensatz zu anderen erneuerbaren Energien, wie der wetterabhängigen Wind- und Sonnenenergie, kann Methan kontinuierlich produziert und gespeichert werden. So lässt es sich je nach Bedarf in Strom oder Wärme umwandeln.
Weitere Informationen im Internet:
www.cebitec.uni-bielefeld.de/grim/index.php/research/metagenomes
(Deutsche Version auswählbar)
In Biogasanlagen arbeiten Heerscharen von Mikroorganismen täglich daran, aus Abfall Energie herzustellen. Um herauszufinden, welche Mikroben diese Arbeit am besten machen, schauen Forscher vom Centrum für Biotechnologie (CeBiTec) der Universität Bielefeld ganz genau hin: Für ausgewählte Biogasanlagen deutschlandweit analysieren sie die Gesamt-Erbinformation der mikrobiellen Gemeinschaften. Die dazu nötigen Sequenzierarbeiten übernimmt ab Anfang 2013 das kalifornische Joint Genome Institute. Am CeBiTec erfolgt die bioinformatische Analyse. Keine einfache Aufgabe, denn die Daten werden in Milliarden von Bruchstücken geliefert, die wiederum von Hunderten von Organismen stammen. Eine Puzzle-Arbeit.
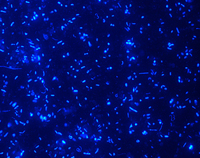
Eingefärbt
fluoreszieren sie unter dem Mikroskop: Die unterschiedlichsten Mikroben
treiben sich in der Probe einer Biogasanlage herum. Welche ihren Job am
besten machen, wollen Forscher der Universität Bielefeld jetzt
herausfinden. Bild: Karsten Niehaus
Erstes Genom entschlüsselt
Einen ersten Erfolg können die Forscher bereits verbuchen. „Die Genomsequenz von einem Methanproduzenten, Methanoculleus bourgensis, konnte am CeBiTec komplett entschlüsselt werden“, berichtet Professor Pühler. Damit wurde in Bielefeld die erste Genomsequenz für ein Methan-produzierendes Archaeon aus einer Biogasanlagen erstellt – ein einzelliges Urbakterium, das in bestimmten Biogas-Anlagen eine wichtige Rolle spielt. Nun wollen die Forscher mehr.
Ortstermin
der Bielefelder Forscher auf dem Gelände einer Bielefelder Biogasanlage
(v.l.): Dr. Andreas Schlüter, Prof. Dr. Jens Stoye, Dr. Alexander
Goesmann, Dr. Alexander Sczyrba und Prof. Dr. Alfred Pühler. Foto: Björn
Fischer
Das Projekt ist Teil des „Community Sequencing Programs“, eines öffentlichen Sequenzierungsprogramms, das am Joint Genome Institute vom amerikanischen Energieministerium finanziert wird. Während bisherige Biogas-Studien sich hauptsächlich auf bestimmte Markergene konzentriert haben, soll jetzt die gesamte Erbinformation der Mikroorganismen untersucht werden. Dazu wird das amerikanische Institut mehr als ein Terabyte an Sequenz-Daten produzieren, was etwa dem Umfang von 300 menschlichen Genomen entspricht. Allerdings werden diese Daten in unzähligen Bruchstücken geliefert. Denn die Millionen von Basen, aus denen ein mikrobielles DNA-Molekül besteht, lassen sich auch mit modernster Technik nicht an einem Stück lesen. Stattdessen liefern die Sequenziertechnologien sich überlappende Abschnitte von etwa 150 Basen - davon aber riesige Mengen. In Milliarden von Bruchstücken werden die DNA-Sequenzen wieder nach Bielefeld geschickt, wo die Arbeitsgruppe „Computational Metagenomics“ (Computergestützte Metagenomik) von Alexander Sczyrba ins Spiel kommt. Sie entwickelt bioinformatische Verfahren zur Rekonstruktion von Genomsequenzen. Ihre Aufgabe ist es, die Daten zu vergleichen, die Überlappungen festzustellen und die Basenabfolge anhand dieser „Klebestellen“ wieder zusammenzusetzen. „Wir versuchen ein Puzzle zu lösen, das aus Milliarden von Teilen besteht, wobei auch noch Hunderte von verschiedenen Puzzle-Spielen miteinander vermischt wurden“, erläutert Sczyrba.
Einzelzell-Genomik verspricht neue Erkenntnisse
Ganz nebenbei werden die Bielefelder Forscher neue Wege in der Genomik beschreiten. Schätzungsweise 99 Prozent aller Mikroorganismen lassen sich im Labor nicht kultivieren. Hier soll eine ganz neue Technologie, die Einzelzell-Genomik, Erkenntnisse bringen, indem die Genomsequenz von einzelnen mikrobiellen Zellen bestimmt wird. Es wird erwartet, dass Kenntnisse über Identität und Funktionen bisher komplett unbekannter Mikroorganismen gewonnen werden können. Im Rahmen des Gemeinschaftsprojekts wird das Joint Genome Institute etwa 100 Einzelzell-Genome sequenzieren.
Rund zwei Jahre haben die Forscher für ihr Projekt eingeplant, an dem auch Bielefelder Doktorandinnen und Doktoranden des Graduiertenclusters in Industrieller Biotechnologie (CLIB) beteiligt sind. Am Ende hoffen sie, die optimale Mikroben-Gemeinschaft für Biogasanlagen zu kennen – und damit dieses Verfahren der Energiegewinnung noch effizienter gestalten zu können.
Hintergrund
Biogasanlagen erzeugen durch die Vergärung von pflanzlicher Biomasse Methan, das für Strom- und Wärmeerzeugung genutzt werden kann. Den Abbau der pflanzlichen Biomasse und die Produktion des Biogases in den landwirtschaftlichen Biogasanlagen wird durch Mikroben bewerkstelligt. Dieser Prozess, der in ähnlicher Form auch im Verdauungstrakt von Rindern abläuft, weist eine ausgeglichene Kohlendioxid-Bilanz auf und trägt damit nicht zum “Treibhauseffekt“ bei. Im Gegensatz zu anderen erneuerbaren Energien, wie der wetterabhängigen Wind- und Sonnenenergie, kann Methan kontinuierlich produziert und gespeichert werden. So lässt es sich je nach Bedarf in Strom oder Wärme umwandeln.
Weitere Informationen im Internet:
www.cebitec.uni-bielefeld.de/grim/index.php/research/metagenomes
(Deutsche Version auswählbar)